Le 4 septembre 2025, la 16e épidémie de MVE depuis 1976 a été déclarée par la République Démocratique du Congo à l’Organisation Mondiale de la Santé. L’épicentre de cette épidémie se situe dans la province du Kasaï – la première résurgence dans cette région depuis 2008. L’analyse génomique de 4 cas positifs a récemment permis de révéler que la souche d’EBOV du Kasaï présenterait une similarité de plus de 99,5 % (seulement 96 mutations) avec la souche responsable de la première épidémie au Zaïre (Yambuku) en 1976. Alors qu’on aurait pu s’attendre à ce qu’EBOV soit proche des souches responsables des épidémies les plus récentes, en raison de son évolution continue, la souche qui a frappé la RDC semble, en partie, inchangée depuis un demi-siècle. Cette proximité entre la souche du Kasaï 2025 avec celle de l’épidémie de 1976 restreint donc les explications plausibles quant au lieu et aux circonstances de la persistance d’EBOV entre les épidémies.
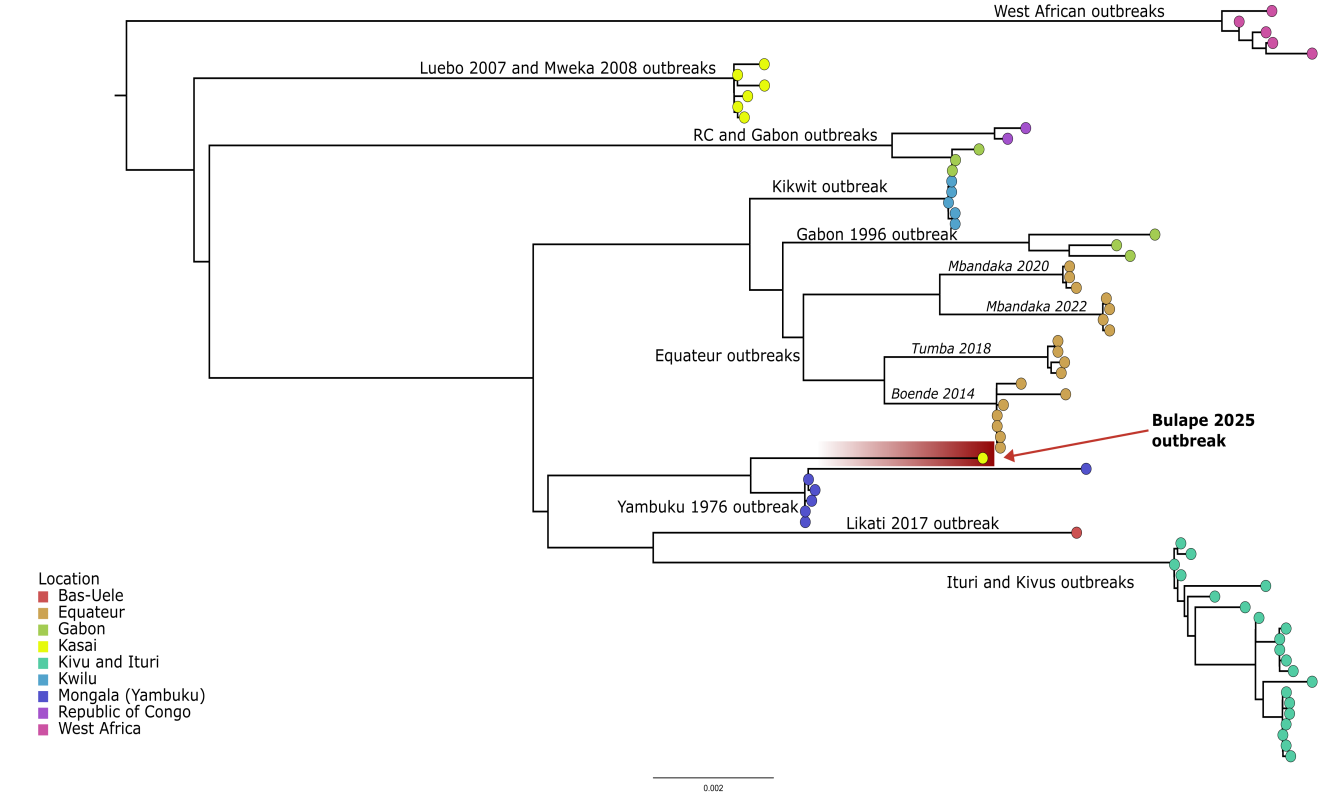
Source : Amuri-Aziza A., Luakanda-Ndelemo G., Makangara-Cigolo J.-C. et al. (2025) The 16th Ebola Virus Disease Outbreak in Bulape Health Zone, Kasai, Democratic Republic of the Congo: A new spillover event from an unknown reservoir host. https://virological.org/t/the-16th-ebola-virus-disease-outbreak-in-bulape-health-zone-kasai-democratic-republic-of-the-congo-a-new-spillover-event-from-an-unknown-reservoir-host/1003/1
Des réservoirs animaux encore mal connus
La question du réservoir d’Ebola — l’hôte animal dans lequel le virus se maintient entre deux flambées humaines — demeure l’un des grands mystères de la virologie tropicale. Depuis 1976, plus de 360 espèces de mammifères et 30 000 individus ont été testés par sérologieSérologie Étude des sérums pour déterminer la présence d’anticorps dirigés contre des antigènes. et PCRPCR "Polymerase Chain Reaction" en anglais ou réaction en chaîne par polymérase en français. Il s'agit d'une méthode de biologie moléculaire d'amplification d'ADN in vitro (concentration et amplification génique par réaction de polymérisation en chaîne), utilisée dans les tests de dépistage. mais des preuves d’infection par l’EBOV n’ont été trouvées que chez 728 individus appartenant à 61 espèces. Des anticorps et des fragments d’ARN ont ainsi été découverts chez les chauves-souris frugivores au Gabon, en République du Congo et au Liberia, mais aucune particule virale infectieuse n’a été isolée à partir de ces espèces potentiellement réservoirs. Il est à noter que d’autres espèces — chimpanzés, gorilles et antilopes céphalophes par exemple — semblent aussi jouer un rôle d’hôtes intermédiaires.
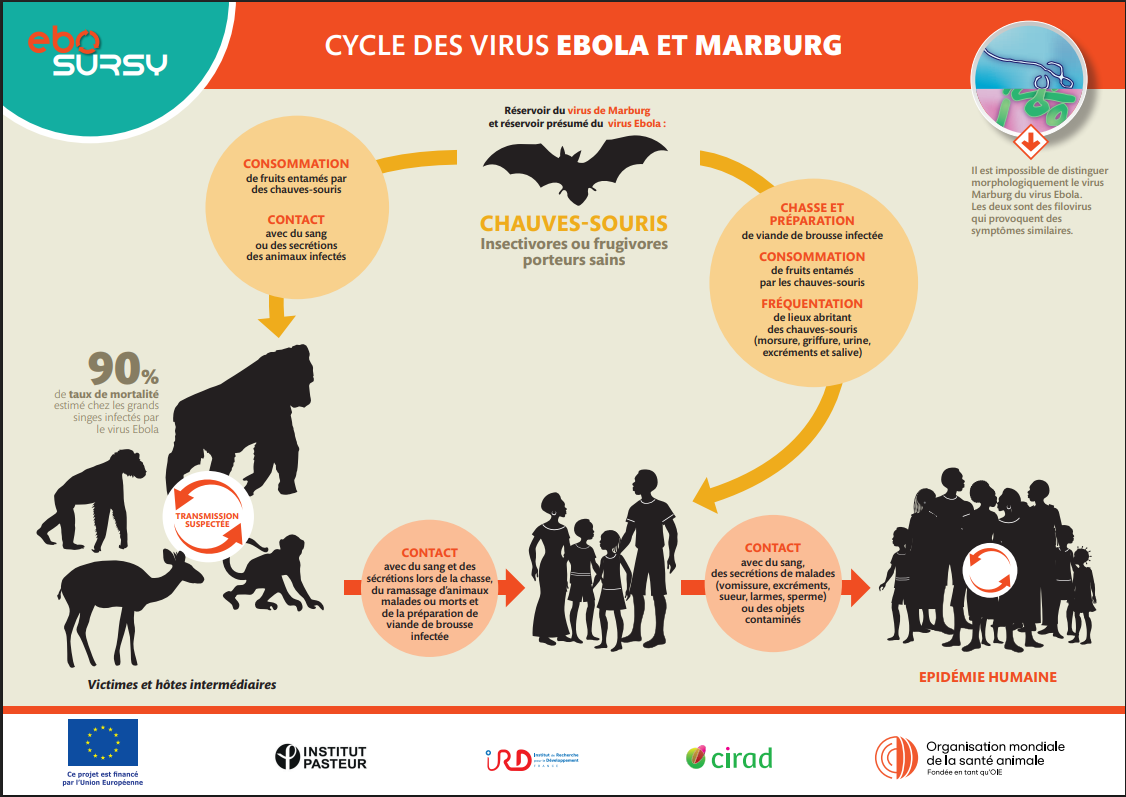
Cycles des virus Ebola et Marbourg. Source : Projet EBO SURSY https://rr-africa.woah.org/app/uploads/2020/08/oi01001_fiche-a4_ebola-marburg.pdf
Étant donné la rareté des infections dans les populations sauvages, la possibilité que l’EBOV soit contenu par un ensemble d’hôtes plutôt que par un seul réservoir majeur reste surprenante – on pourrait en effet s’attendre à des infections chez les différentes espèces.
D’après des études antérieures, l’absence d’infections documentées reflète un échantillonnage insuffisant des espèces et des populations.
Autre explication possible : la dynamique de l’EBOV et du système immunitaire au sein de l’hôte est plus complexe qu’on ne l’avait supposé jusqu’ici. D’importantes espèces réservoirs peuvent ne pas avoir été détectées jusqu’à présent, si l’échantillonnage a été effectué au mauvais moment et au mauvais endroit, ou si le virus était dormant dans des tissus non testés.
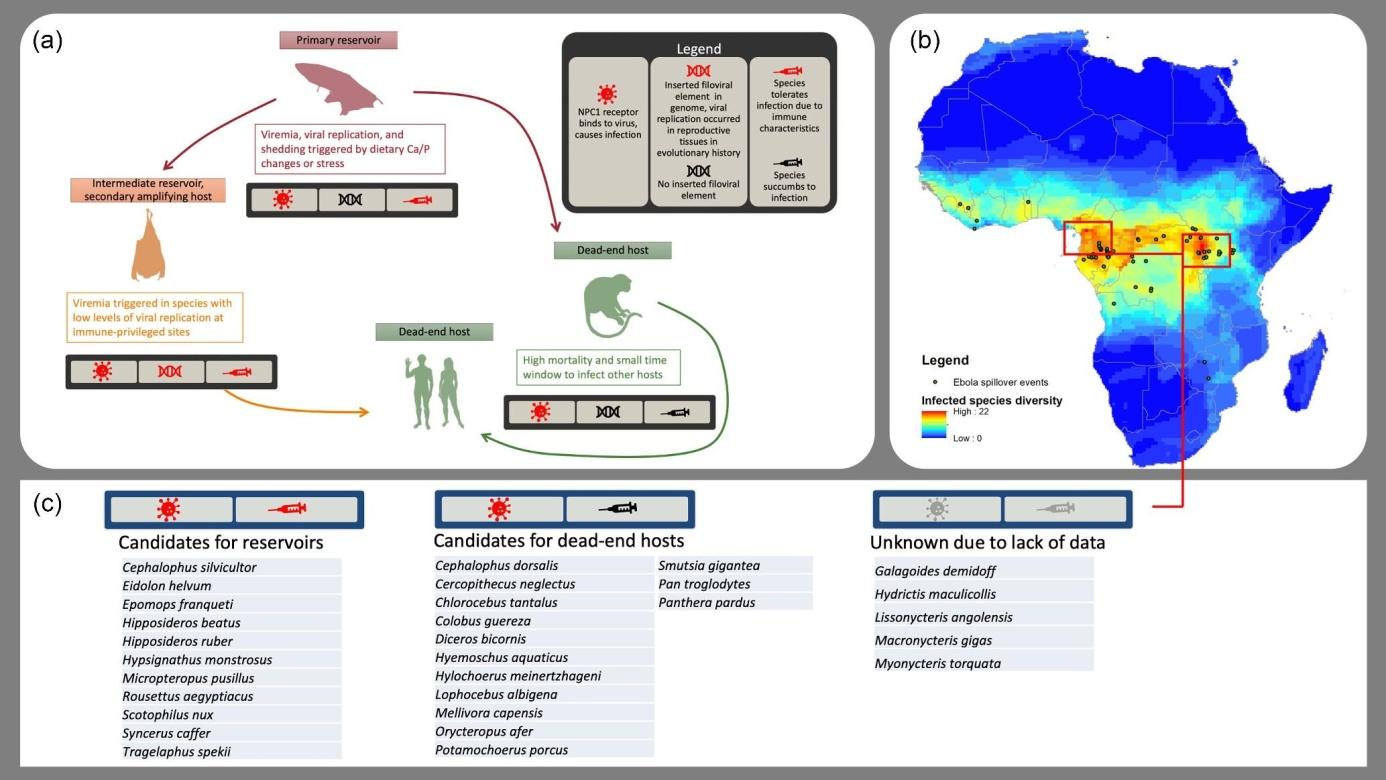
Hôtes et types de réservoirs d’EBOV. Le panneau (a) présente 3 types d’hôtes du virus Ebola selon leur mode de transmission virale et leur dynamique évolutive, ainsi que les méthodes pour les identifier. Le panneau (b) illustre la richesse des espèces de mammifères infectées par le virus Ebola et les sites de transmission zoonotique connus jusqu’en 2021. Les zones encadrées sur la carte mettent en évidence les régions présentant la plus forte richesse d’espèces connues pour être positives aux anticorps ou par PCR. Enfin, le panneau (c) fournit des listes d’espèces réservoirs putatifs, d’hôtes accidentels et d’espèces pour lesquelles les données sont insuffisantes (c’est-à-dire des espèces à fort potentiel de réservoir qui n’ont jamais fait l’objet d’un échantillonnage pour la recherche d’anticorps ou par PCR) dans les régions d’Afrique présentant la plus forte richesse en espèces infectées. Les prédictions concernant les réservoirs et les hôtes accidentels sont basées sur des modèles développés précédemment. Source Mekala Sundaram et al. (2025) https://doi.org/10.1093/biosci/biaf050
Le modèle SILI (voir figure ci-après) décrit les variations temporelles des titres en virus et en anticorps anti-EBOV chez les hôtes et réservoirs potentiels, avec les mécanismes sous-jacents aux pics de réplication virale. Ce modèle propose aussi une stratégie d’échantillonnage théorique pour identifier les espèces animales réservoirs d’EBOV.
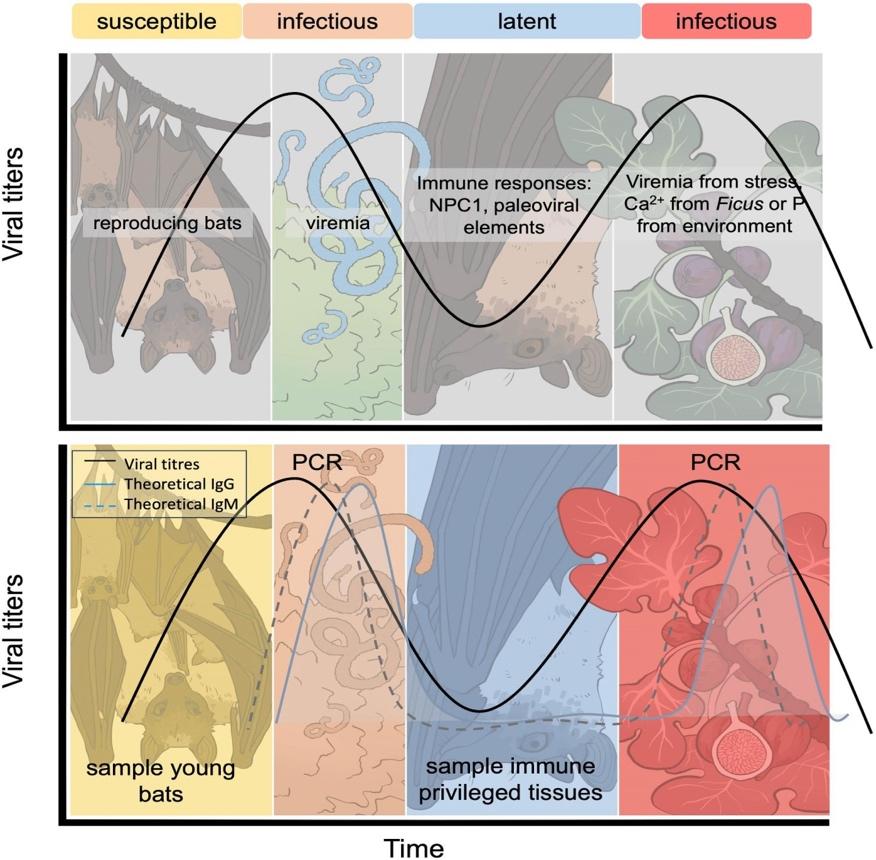
Le modèle SILI (Susceptible-Infectious-Latent-Infectious) Le schéma supérieur illustre les mécanismes sous-jacents aux pics de réplication virale. Le schéma inférieur propose une stratégie d’échantillonnage pour identifier un réservoir du virus Ebola. Les courbes théoriques des IgG et IgM sont basées sur les résultats d’une équipe. Cependant, les variations temporelles naturelles des titres d’anticorps n’ont pas été quantifiées chez les chauves-souris. Source : Mekala Sundaram et al. (2025) https://doi.org/10.1093/biosci/biaf050
Une persistance virale chez l’hôte qui surprend
Une autre hypothèse avancée est celle de la persistance : l’EBOV pourrait « hiberner » dans les tissus de son hôte, alternant entre phases de réplication et périodes de dormance. Ce scénario a notamment été envisagé lors de l’épidémie de MVE en Guinée en 2021, survenue plusieurs années après la grande épidémie de 2013-2016 en Afrique de l’Ouest. La souche d’EBOV identifiée en 2021 présente une divergence génétique bien moindre que ce à quoi on pourrait s’attendre dans le cadre d’une transmission interhumaine continue, ce qui laisse supposer une infection persistante avec une faible réplication ou une longue latence — un phénomène qui n’a jamais été observé chez les animaux sauvages. Les séquences génétiques de la souche Kasaï 2025 suggèrent une fois encore que l’EBOV pourrait avoir persisté de la même manière chez l’humain.
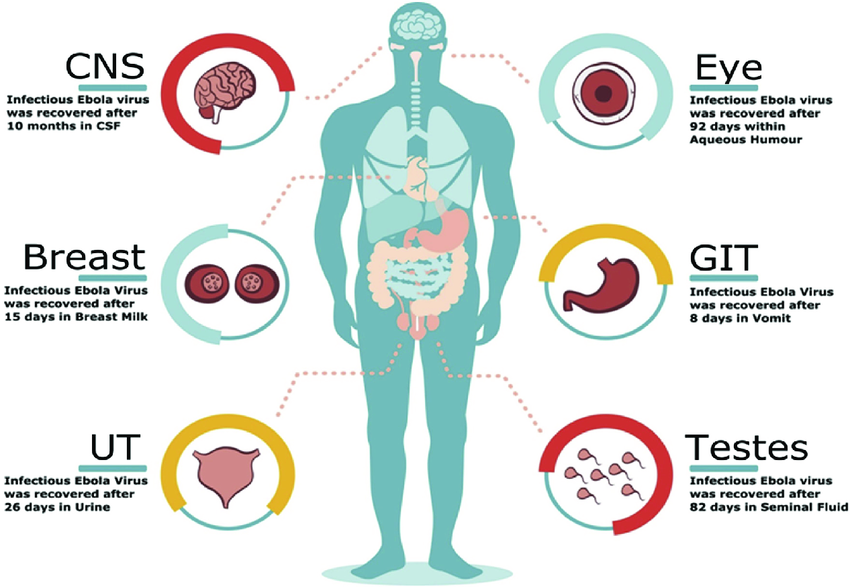
Données sur la persistance du virus Ebola dans différents fluides corporels ou tissus après l’apparition de la maladie. Des particules virales infectieuses d’EBOV ont été identifiées dans les fluides ou tissus corporels des survivants : liquide céphalo-rachidien, lait maternel, liquide séminal, sécrétions vaginales, prélèvements gastro-intestinaux (écouvillons rectaux, selles, salive, vomissements), urine, voies respiratoires inférieures (alvéoles), yeux (humeur aqueuse, larmes, conjonctives) et autres (peau, sueur, placenta, sang du cordon ombilical et liquide amniotique) pendant des périodes prolongées après l’apparition de la MVE. Source :Haaris A. et al. (2017), https://doi.org/10.1016/j.apjtm.2016.12.008
Jusqu’à 40 % des personnes ayant survécu à une infection par l’EBOV développent une inflammation récurrente dans certains organes ou tissus (œil, testicule, synovie, méninges), malgré des tests virologiques négatifs. Ces sites présentent souvent une immunité atténuée (ou un privilège immunitaire). Lors des récidives (uvéite, synovite, arthrite…), l’ARN d’EBOV est fréquemment détecté dans le compartiment extracellulaire et le virus peut être facilement cultivé à partir d’échantillons de ces tissus, ce qui suggère qu’une infection persistante ou latente est à l’origine de l’inflammation. Dans le cas de MVE, on présume que la récidive est due à une infection persistante chronique plutôt qu’à une réactivation d’une infection latente, comme c’est le cas pour les virus à ADN tels que les herpès virus.
- Dans l’infection persistante chronique, la maladie est causée par un virus en réplication active, à un faible niveau.
- En revanche, dans l’infection latente, la réplication a cessé, bien que le virus reste capable de se répliquer.
Cette distinction est importante car la transmission de l’EBOV se fait par contact direct. La présence d’un virus capable de se répliquer, associée à la durée de l’infection, a donc des conséquences sur la contagiosité, notamment chez les professionnels de santé prenant en charge des personnes guéries de MVE.
Quid de la latence ?
Les données semblent contradictoires à ce stade.
D’un point de vue virologique, pour déterminer si les infections persistantes sont caractérisées par une réplication virale continue, des scientifiques ont séquencé l’EBOV prélevé dans le sperme d’hommes ayant survécu à une MVE et dans un groupe en phase aiguë. Résultat, les taux d’évolution de l’EBOV (défini par le taux de mutation, autrement dit le nombre de substitutions/site/an) pendant la phase de persistance étaient similaires ou inférieurs à ceux observés lors de la phase aiguë. La réplication/transcription active de l’EBOV s’est poursuivie pendant la convalescence, mais a diminué au fil du temps. Les auteurs pointent une sélection naturelle éliminant les mutations défavorables, ce qui est compatible avec une persistance virale plutôt qu’une latence. Enfin, il ne semble pas que l’EBOV ait subi des pressions de sélection sensiblement différentes lors de sa persistance dans les organes et tissus à privilège immunitaire (testicule, œil) par rapport à celles subies lors des infections aiguës. Cependant, un relâchement modéré des contraintes de sélection pour une protéine virale a été observé pendant la persistance, ce qui semble indiquer que la persistance de l’EBOV dans le sperme, l’urine et l’humeur aqueuse ne correspond pas à une infection quiescente ou latente.
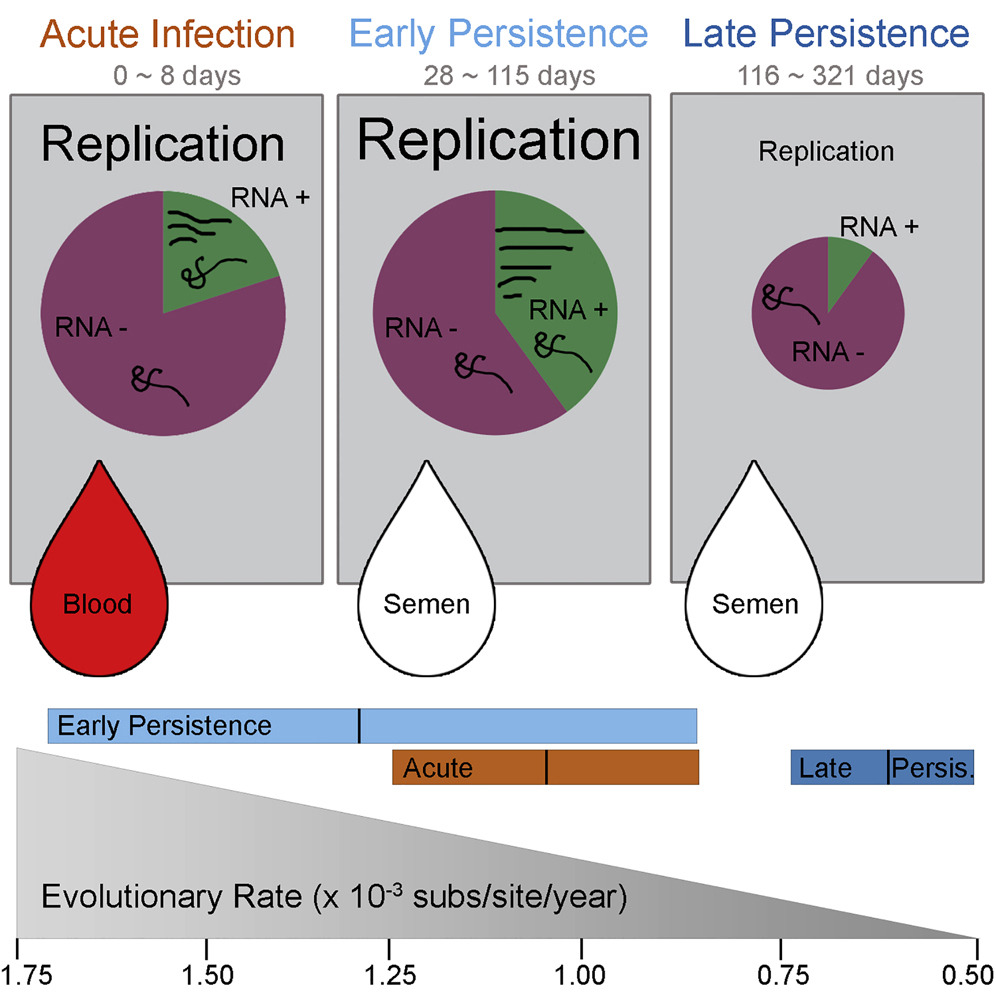
Réplication active du virus Ebola, y compris pendant la persistance, et variation du taux d’évolution chez les survivants de la MVE. Source Whitmer S.L.M et al; (2018) https://doi.org/10.1016/j.celrep.2018.01.008
Mais d’un point de vue épidémiologique, un preprint (octobre 2025) récent d’une équipe internationale dirigée par Andrew Rambault semble pencher vers l’hypothèse de la latence pour expliquer les épidémies d’Ebola : si la communauté scientifique pensait initialement que toutes les souches d’EBOV descendaient d’une même origine apparue dans les années 1970, l’épidémie d’Afrique de l’Ouest en 2013 a remis cette idée en question, car elle était géographiquement très éloignée et génétiquement un peu différente ; de nouvelles analyses ont en outre montré que le rythme d’évolution d’EBOV était surestimé, avec certaines branches épidémiologiques mal interprétées. Des études récentes ont aussi révélé que le virus Ebola peut rester « endormi » pendant de longues périodes (comme en Guinée, voir plus avant). Cette découverte suggère que le virus pourrait aussi rester « latent » chez les animaux, ce qui expliquerait pourquoi certaines épidémies récentes montrent peu de différences génétiques avec les anciennes.
En s’appuyant sur un modèle de latence évolutive inspiré des observations de virus persistants chez des survivants d’Ébola, les chercheurs ont calculé que l’EBOV existerait depuis au moins les années 1920–1930. Pendant son évolution, le virus alternerait entre des phases de multiplication active et des phases de dormance. Ces périodes d’inactivité pourraient durer des dizaines d’années, ralentissant ainsi son évolution apparente. En appliquant ce modèle à 19 séquences représentatives d’épidémies indépendantes, ils ont découvert que les estimations classiques surestimaient d’un facteur deux à quatre la vitesse d’évolution d’Ebola. Ce modèle expliquerait mieux les différences entre les épidémies et les longues périodes sans flambées épidémiques.
Comprendre que le virus peut rester caché longtemps chez différents hôtes change la façon de le surveiller. Certaines épidémies récentes qu’on croyait liées à des survivants humains pourraient en réalité être dues à des EBOV restés dormants chez des animaux. Cela montre aussi qu’EBOV circule depuis beaucoup plus longtemps qu’on ne le pensait. Le phénomène de latence pourrait permettre au virus de survivre dans les colonies de chauves-souris et de réapparaître des années plus tard. Une découverte qui pourrait aider à prévoir et à prévenir les futures flambées.
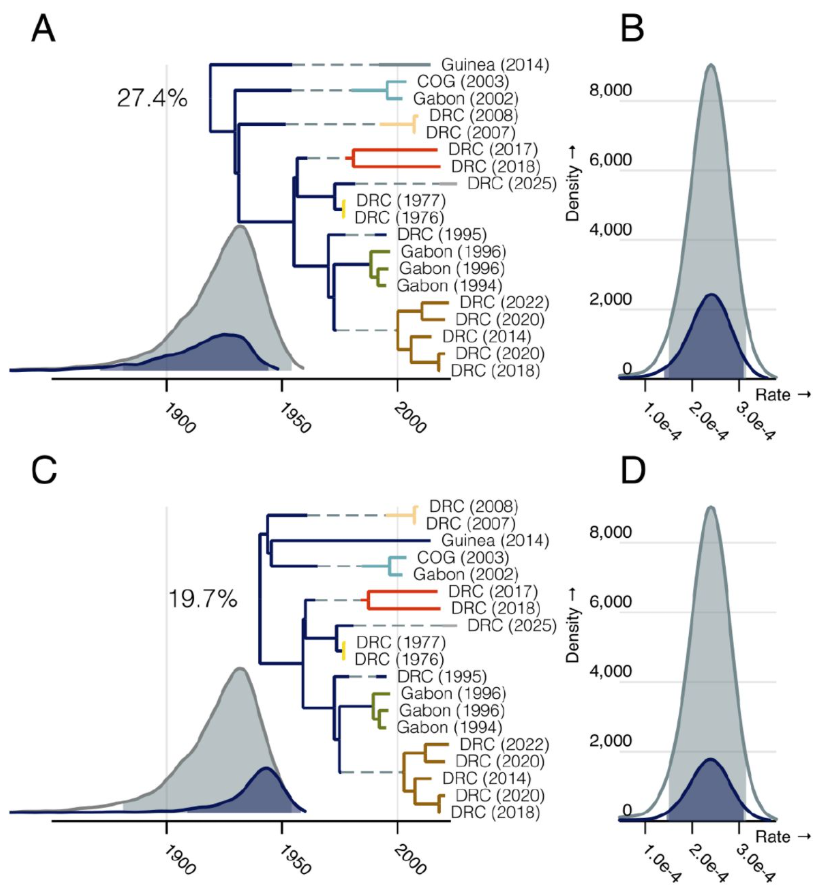
Estimations du modèle de latence du virus Ebola selon deux hypothèses différentes quant à la position de la racine de l’arbre évolutif avec la distribution du taux d’évolution du virus pendant les phases actives de réplication. La durée moyenne de latence (conditionnée par la présence d’au moins une période de latence) est représentée par une ligne pointillée sur les branches dont la probabilité a posteriori d’une latence quelconque est supérieure à 50 %. Source : McCrone J.T., et al. (2025) https://doi.org/10.1101/2025.10.17.683
Une circulation lente à bas bruit…
Une autre explication serait une circulation virale lente associée à une très faible prévalencePrévalence Nombre de personnes atteintes par une infection ou autre maladie donnée dans une population déterminée. Dans ce cas, le virus ne disparaît jamais complètement, mais se transmettrait si rarement que des chaînes de transmission minimales subsisteraient. Avec si peu d’occasions de se répliquer, il accumulerait des mutations très lentement, comme le virus Marburg chez les chauves-souris frugivores. Cependant, l’extraordinaire stabilité de la souche du Kasaï 2025 met à mal ce modèle.
… ou un savant équilibre écologique ?
Enfin, une dernière possibilité serait une forme d’équilibre écologique, dans lequel EBOV se répliquerait continuellement chez des hôtes dont le système immunitaire serait capable de contrôler la réplication sans éliminer complètement le virus.. Des mutations apparaitraient mais persisteraient rarement, créant une apparence de stabilité. Cette explication semble moins convaincante, car elle nécessiterait un hôte réservoir stable et une élimination constante des mutations, ce qui reste à prouver.
Chaque explication prédit des schémas distincts : la persistance produirait des génomes quasi identiques à partir des mêmes populations hôtes à des années d’intervalle ; une circulation lente montrerait une dérive génétique progressive ; et l’équilibre révélerait un virus en continue avec une diversité limitée. L’obstacle ne réside pas dans le manque d’hypothèses, mais dans l’absence de travaux de terrain écologiques systématiques et à long terme pour les tester.
Conclusion
L’épidémie de MVE du Kasaï cette année semble renforcer l’hypothèse selon laquelle la stase génomique pourrait être une caractéristique récurrente de l’écologie d’EBOV. Une lignée pratiquement inchangée depuis 1976 remet en question les hypothèses sur l’évolution d’EBOV et met en évidence les angles morts de la surveillance. Les succès obtenus avec les virus Marburg et Bombali montrent qu’une approche « One Health », qui consiste à surveiller conjointement les survivants humains, la faune sauvage et les changements écologiques, offre les meilleures chances de détecter la persistance avant la transmission inter-espèces. Tant que ces mécanismes ne seront pas compris, les contre-mesures resteront réactives et l’EBOV continuera de réapparaître sous forme de flambées épidémiques de MVE.
Références
- Amuri-Aziza, A ∙ Luakanda-Ndelemo, G ∙ Makangara-Cigolo, J-C ∙ et al. (2025) The 16th Ebola virus disease outbreak in Bulape Health Zone, Kasai, Democratic Republic of the Congo: a new spillover event from an unknown reservoir host. Virological.org https://virological.org/t/the-16th-ebola-virus-disease-outbreak-in-bulape-health-zone-kasai-democratic-republic-of-the-congo-a-new-spillover-event-from-an-unknown-reservoir-host/1003/1 Date de publication : le 04 septembre 2025, accédé le 11 novembre 2025
- Shiwani H. A., et al. (2017) , An update on the 2014 Ebola outbreak in Western Africa, Asian Pacific Journal of Tropical Medicine, Volume 10, Issue 1, 2017, Pages 6-10, https://doi.org/10.1016/j.apjtm.2016.12.008 https://www.sciencedirect.com/science/article/pii/S1995764516307386
- McCrone J.T., et al. (2025) Evidence of latency reshapes our understanding of Ebola virus reservoir dynamics, (2025), PREPRINT, bioRxiv 2025.10.17.683141; https://doi.org/10.1101/2025.10.17.683
- Leroy E.M., Gonzalez J-P, Baize S., Ebola and Marburg haemorrhagic fever viruses: major scientific advances, but a relatively minor public health threat for Africa, Clinical Microbiology and Infection, 17(7), 2011, 964-976, https://doi.org/10.1111/j.1469-0691.2011.03535.x, https://www.sciencedirect.com/science/article/pii/S1198743X14613744
- Mekala Sundaram, Nicole L Gottdenker, Patrick R Stephens (2025) Where is the elusive primary ebolavirus resevoir and how do we find It?, BioScience, 2025, biaf050, https://doi.org/10.1093/biosci/biaf050
- Pirofski L-A, Casadevall A. (2020), The state of latency in microbial pathogenesis J Clin Invest. 2020;130(9):4525-4531. https://doi.org/10.1172/JCI136221
- Projet EBO SURSY (https://ebo-sursy.woah.org/fr/)
- Whitmer S.L.M., et al. (2018), Active Ebola Virus Replication and Heterogeneous Evolutionary Rates in EVD Survivors, Cell Reports, Volume 22, Issue 5, 2018, Pages 1159-1168. https://doi.org/10.1016/j.celrep.2018.01.008
- WHO – Ebola virus disease—Democratic Republic of the Congo (2025) https://www.who.int/emergencies/disease-outbreak-news/item/2025-DON580 Date de publication : le 05 septembre 2025, accédé le 08 septembre 2025
